Algoritmo desarrollado en la UTEM optimiza predicción del metabolismo celular
Autor: Joaquín|
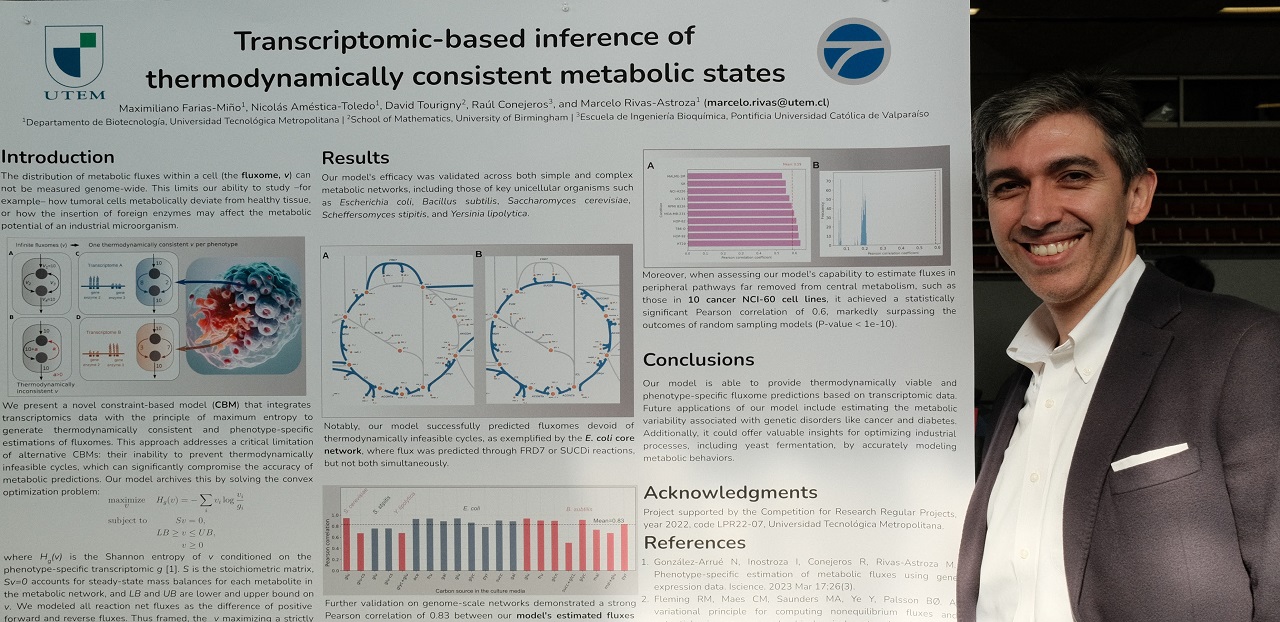
La comunidad científica fue testigo de un gran avance científico en la Annual International Conference on Research in Computational Molecular Biology (RECOMB 2024) realizada en el Massachusetts Institute of Technology (MIT) durante este 29 de abril y 2 de mayo. Fue en este escenario donde Marcelo Rivas Astroza, académico del Depto. de Biotecnología de la FCNMMA y jefe del Programa de Biología Computacional, presentó el algoritmo Pheflux-TIC, capaz de realizar predicciones del metabolismo celular en tan sólo 3 minutos, un proceso que anteriormente tomaba hasta una semana.
PheFlux-TIC es una extensión de PheFlux, algoritmo que permite inferir el metabolismo celular condicionado en datos fenotipo-específicos de expresión genética. Rivas Astroza explica que “El algoritmo que hemos desarrollado predice el estado metabólico de una célula, es decir, el conjunto de reacciones químicas que ocurren dentro de ella para obtener energía y componentes básicos”.
“Lo innovador de nuestro algoritmo es que, a diferencia de otros modelos, tiene en cuenta las leyes de la termodinámica, asegurando que las predicciones sean consistentes con los principios fundamentales de la física que rigen estos procesos. Esto es crucial porque los modelos anteriores a menudo predecían estados metabólicos imposibles desde el punto de vista termodinámico”, enfatiza el académico.
El proyecto, surge de una colaboración entre la UTEM, la Universidad de Birmingham y la Pontificia Universidad Católica de Valparaíso (PUCV), e involucra a Rivas Astroza, junto a los estudiantes Maximiliano Farías Miño y Nicolás Améstica Toledo, y a los profesores David Tourigny y Raúl Conejeros Risco, de la Universidad de Birmingham y la Pontificia Universidad Católica de Valparaíso. “Maximiliano y Nicolás han desempeñado un papel fundamental en el desarrollo e implementación del algoritmo. Sus contribuciones han sido esenciales para el éxito del proyecto, incluyendo el desarrollo del código computacional y la validación de las predicciones del modelo”, destaca Rivas Astroza.

Avance científico pionero
PheFlux-TIC surgió de la necesidad de mejorar la precisión de los modelos existentes para predecir el metabolismo celular. “En la actualidad, estos modelos generan artefactos matemáticos conocidos como ciclos termodinámicamente infactibles (CTI), análogos a intentar usar una bicicleta perpetuamente en una colina sin pedalear, lo cual es imposible en la realidad y no obedece las leyes termodinámicas que rigen el universo. Estos ciclos infactibles pueden comprometer la relevancia biológica y al precisión predictiva del modelo, por lo que es esencial diseñar herramientas computacionales más sofisticadas que puedan diferenciar entre rutas metabólicas termodinámicamente factibles o infactibles”, explica Nicolás Améstica Toledo.
Las expectativas para el algoritmo Pheflux-TIC son altas. En palabras de Rivas Astroza: “Esperamos que nuestro algoritmo se convierta en una herramienta estándar en el campo de la biología de sistemas y la biotecnología. Creemos que puede ser útil para estudiar enfermedades como el cáncer y la diabetes, así como para optimizar procesos industriales como la fermentación”.

La relevancia de este desarrollo radica en su potencial para mejorar la comprensión de los procesos biológicos fundamentales y para desarrollar nuevas terapias y biotecnologías. “Al predecir con mayor precisión el metabolismo celular, podríamos identificar nuevas dianas terapéuticas para enfermedades, diseñar microorganismos más eficientes para la producción de biocombustibles y otros productos, y personalizar tratamientos médicos en función del perfil metabólico de cada paciente”, comenta Marcelo Rivas Astroza.
Este avance no sólo representa un salto significativo en la biología computacional, sino que también pone de relieve la capacidad innovadora de los investigadores de la UTEM, en conjunto con otros expertos, tanto en el ámbito científico nacional e internacional. “Nuestros próximos pasos involucran la producción de un artículo para su publicación en una revista científica”, expresa Améstica Toledo.